E. coli (EC) – coltivazione e sierotipizzazione
E. coli è un batterio gram-negativo a forma di bacillo che ha innumerevoli varianti. Oltre ai ceppi commensali, che in modo del tutto naturale si trovano nella flora intestinale di tutti i mammiferi, ci sono ceppi classificati come E. coli intestinale (InPEC) o extraintestinale (ExPEC). Senza dubbio, la differenza tra queste varianti è stata a lungo un problema per la diagnostica, dato che differenti patotipi non potevano essere identificati con metodi fenotipici semplici.

Gli E. coli possono essere facilmente identificati attraverso una coltura in agar-sangue (figura 1) o terreni differenziali (p. es., agar Gassner o MacConkey) di campioni adeguati (intestino piccolo, feci) di suini con infezione acuta. Anni fa, era stata utilizzata con successo la sierotipizzazione per differenziare gli isolati di E. coli, dato che certi sierotipi sono frequentemente associati a malattie specifiche (tabella 1). Pertanto, in veterinaria, è routine determinare gli antigeni O e F, per agglutinazione rapida su piastra utilizzando sieri poli o monovalenti (gli antigeni O sono componenti degli LPS della parete cellulare batterica e sono anche endotossici; gli antigeni F [fimbriali] sono adesine che facilitano l'adesione ospite-specifico per colonizzare le cellule dell'intestino piccolo). Nonostante questo metodo sia rapido, è soggetto ad errori per reazioni crociate, mascheramento degli antigeni diana o per l'incapacità delle fimbrie a svilupparsi in vitro, il che può condurre ad una classificazione erronea. Da un altro lato, da decadi la sieroformula O:K:H viene e continua ad essere lo standard per la determinazione completa di tutti i sierotipi, per esempio, per determinare non solo gli antigeni O, ma anche quelli K (polissacaride capsulare) e H (proteina flagellare).
Tabella 1: Patogeni E. coli intestinali nei suini
| Patotipi |
Malattie causate da E. coli |
Sierotipi con antegene O frequentemente associati | Sierotipi fimbriali frequentemente associati |
| E. coli patogeno intestinale (InPEC) | Diarrea da E. coli nei suinetti sottoscrofa | O8, O108, O138, O139, O141, O147, O149 e O157 | F4, F5, F6 e F41 |
| Diarrea da E. coli nei suinetti svezzati | O8, O108, O138, O139, O141, O147, O149 e O157 | F4, F18ac | |
| Malattia degli edemi in suinetti svezzati | O138, O139 e O141 (O147, O157 e routinariamente non sierotipizzabili) | F18ab |
E. coli (EC) – patotipi e "virotipizzazione"
Altri progressi nella metodoloiga biomolecolare e miglioramenti delle analisi biologiche molecolari nei riguardi dell'E. coli, hanno portato all'identificazione di molti geni che codificano i fattori di virulenza (tabella 2): da una parte, i fattori classici di virulenza con effetti nocivi (tossine) e dall'altra quelli che direttamente o indirettamente facilitano la colonizzazione e la sopravvivenza dell'E. coli nell'ospite. L'identificazione di questi geni, per esempio, tramite PCR o tecniche di array, rende possibile la determinazione del potenziale di virulenza degli isolati di E. coli e di distinguire tra differenti patotipi (tabella 3). L'identificazione di questi marcatori di virulenza, e pertanto dei suoi geni diana, si chiama "virutipizzazione".
Tabella 2: Esempi di geni diana rilevati negli E. coli ed i fattori di virulenza
(fimbrie, adesine, tossine) e la loro codifica
| Geni diana | Fattori di patogenicità |
| faeG (F4) | Fimbrie F4 |
| fanC (F5) | Fimbrie F5 |
| fasA (F6) | Fimbrie F6 |
| fedA (F18) | Fimbrie F18 |
| fim41A (F41) | Fimbrie F18 |
| fimH (F1) | Fimbrie tipo 1 |
| fimA (F1) | Fimbrie tipo 1 |
| papC | Fimbrie P |
| aidA (AIDA) | Adesina autotrasportatrice AIDA-I |
| paa | Fattore di aderenza suina |
| eaeA (intimina) | Intimina |
| eltB (LTI) | Enterotossina sensibile al caldo |
| estA (STI) | Enterotossina resistente al caldo |
| est B (STII) | Enterotossina resistente al caldo |
| estA (EAST) | Enterotossina resistente al caldo |
| stx2e | Shigatoxina variante 2e |
| cdtB | Tossina citoletale distending |
| cnf1 | fattore citotossico necrotizzante tipo 1 |
| iucD | Aerobactina |
| escV | Sistema di secrezione tipo III |
| pic | Autotrasportatore serin proteasi |
Tabella 3: Geni diana per l'identificazione dei patotipi di E. coli nei suini
| Patotipi | Fimbrie e adesine (geni diana) | Tossine (geni diana) | Sintomi | |
| E. coli patogeno intestinale (InPEC) |
E. coli enteropatogenico (EPEC)
|
fimA/fimH paa intimina |
escV pic cdtB |
diarrea da malassorbimento |
|
E. coli enterotossico (ETEC)  |
F4, F5, F6, F18, F41 fimA/fimH AIDA paa |
STI, STII, EAST LTI Stx2e emolisina |
diarrea da ipersecrezione | |
| Malattie degli edemi E. coli (EDEC) | F18 AIDA |
Stx2e emolisina |
malattia degli edemi | |
| E. coli produttore di Shigatoxina (STEC) | fimA, fimH intimina |
Stx2e EAST escV pic, cdtB emolisina |
diarrea catarrale-emorragica | |
| E. coli patogeno extraintestinale(ExPEC) |
E. coli setticemica (SEPEC) |
papC | iucD | setticemia emorragica |
| E. coli uropatogenica (UPEC) | papC fimA, fimH | cnf1, iucD emolisina |
mastiti da coliformi nelle scrofe | |
Tutti gli isolati di E. coli patogeni contengono sempre una combinazione di fimbrie ed adesine,oltre che tossine. Varie tossine ed adesine possono essere presenti assieme e con differenti combinazioni. La determinazione di questi vari marcatori di virulenza e l'identificazione dei geni, offre informazioni sopratutto sul patotipo di E. coli isolato nella coltura cellulare (tabella 3). E' necessario evidenziare che gli E. coli emolitici non sono gli unici che sono patogeni (figura 1), e difatti, gli E. coli non emolitici, in particolare, sono diagnosticati con frequenza come causa di malattia clinica. Gli E. coli emolitici normalmente non possiedono geni rilevanti per le fimbrie o tossine. Invece, se è possibile, tutte le varianti degli isolati di E. coli identificate tramite colture, devono essere virotipizzate con la finalità di assicurare che il vero patogeno responsabile possa essere identificato. Le informazioni sui tipi e tossine fimbriali di E. coli patogeno identificati, sono utili anche per la selezione di vaccini adeguati per gli antigeni corrispondenti.
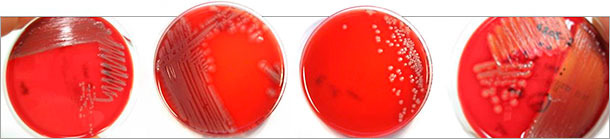
Figura 1: Differenti morfologie delle colonie degli isolati di E. coli patogeni in agar sangue (Columbia) identificati dopo la virotipizzazione con PCR multiplae. Non solamente gli E. coli emolitici (destra) sono patogeni.
Nell'interpretazione dei risultati bisogna tenere in considerazione che i patotipi di E. Coli possono essere isolati da ospiti sani (infezione latente), sopratutto della flora intestinale. Quindi la valutazione dei risultati può solamente essere fatta considerando la storia clinica del suino, così come la concentrazione di patogeni isolati assieme al patotipo identificato.
L'interpretazione del risultato della virotipizzazione diventa complicata quando i marcatori di virulenza vengono identificati direttamente nel campione realizzato, per esempio, nelle feci, senza la tipizzazione dei singoli isolati. Anche se tecnicamente possibile, viene realizzato sotto richiesta, quando differenti patotipi di E. coli sono presenti nel campione in esame, si ottiene anche un mix di tutti i fattori rilevabili, portando all'identificazione di falsi patotipi ed ad una combinazione dei marcatori di virulenza che senza questo non ci sarebbero. Inoltre, è possibile che si identifichino marcatori di virulenza di altri batteri intestinali presenti nei campioni fecali, dato che questi batteri hanno geni similari. In questo caso, è possibile che si rilevino marcatori di virulenza per gli E. coli, e che di fatto non sono i fattori di virulenza dell'E. coli identificato nel campione stesso. Per cui è necessario fare attenzione nell'interpretazione dei risultati ottenuti nella virotipizzazione dei campioni.
In combinazione con l'esame clinico è spesso necessario avere l'esame patologico (descritto in precedenza) e con diagnosi differenziali adeguate: la virotipizzazione degli isolati di E. coli offre un miglioramento importante nella diagnostica delle infezioni da E. coli nel suino.



