Rilevazione degli antigeni virali della PRRS
Una diagnosi definitiva della PRRS di suini ammalati può essere realizzata con la rilevazione delle lesioni microscopiche caratteristiche del PRRSv assieme alla dimostrazione dell'antigeno virale nei tessuti lesionati. Per identificare l'antigene (i) del virus PRRS nei tessuti, si può utilizzare l'immunofluorescenza (IF) oppure l'immunoistochimica (IHC) da sezioni di tessuti congelati (figura 1). Questi tests si combinano con gli anticorpi monoclonali o policlonali specifici per il PRRSv. Il test di IF diretto su sezioni di tessuti congelati è economico e veloce. E' molto specifico, ma non sempre molto sensibile. I risultati vengono particolarmente influenzati dalla qualità del tessuto(p. es. autolisi). In cambio, l'IHC è utile per identificare l'antigene virale nei tessuti fissati con formalina. L'IHC è più sensibile rispetto alla IF diretta, ma impiega più tempo ed è più costosa.

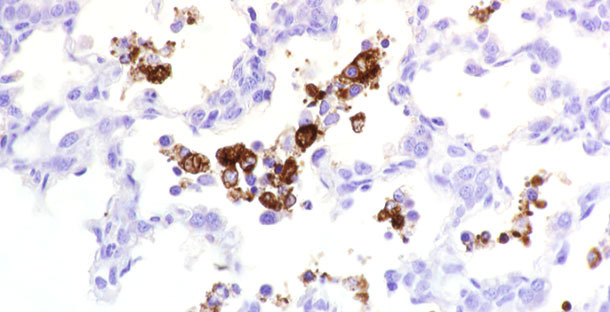
Figura 1. Antigeno di PRRSv rilevato su tessuto.
Per la IF diretta si devono inviare tessuti freschi o congelati e se si desidera l'IHC, devono essere fissati in formalina neutra tamponata al 10%. Senza dubbio, la fissazione prolungata dei tessuti in formalina è dannosa per l'identificazione degli antigeni medianti l'IHC. Se si prevede un ritardo nella realizzazione del test, si consiglia di sottoporre i tessuti in alcool dopo la fissazione con la formalina. I tessuti migliori per questi tests sono: cuore, reni, polmoni, linfonodi, milza, timo e tonsille. Gli antigeni del virus PRRS possono essere ugualmente rilevati nelle ghiandole surrenali, intestino, fegato ed a volte, cervello. Nel realizzare l'IF e l'IHC, si possono utilizzare anticorpii monoclonali specifici per gli antigeni altamente conservati tra i ceppi isolati degli USA ed Europa, oppure si possono utilizzare anticorpi monoclonali contro epitopi meno conservati per valutare le differenze antigeniche tra i ceppi. Se la diagnosi di PRRS viene realizzata con IF o IHC, a causa dell'elevata specificità degli anticorpi monoclonali e della variabilità antigenica del virus PRRS, può essere indicato usare più di un anticorpo monoclonale per evitare una diagnosi sbagliata.
Rilevazione di materiale genomico virale della PRRS
Sono stati sviluppate tests basati sulla reazione a catena della polimerasi (PCR) per identificare l'RNA del PRRSv (ossia, acido nucleico) in campioni di campo. Dato che il virus non ha bisogno di essere isolato in coltivazione cellulare affinchè l'RNA venga identificato, la PCR può offrire in modo più veloce il risultato del test per l'RNA virale. Come principio generale, si considera che i tests realizzati con PCR sono molto sensibili e molto specifici.
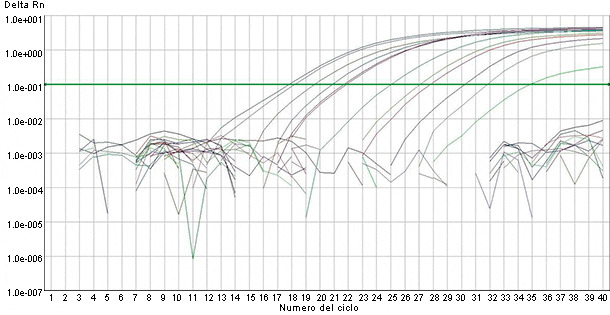
Figura 2. Rilevazione del materiale genomico del virus PRRS in campioni di campo mediante PCR fluorogenica automatizzata.
Sono stati creati vari tipi di tests PCR che usano primers specifici complementari alla sequenza dei differenti frammenti genomici per l'identificazione di materiali genetici virali della PRRS nei campioni clinici. In qualsiasi analisi che usa la PCR per la PRRS, l'RNA virale deve essere estratto dai campioni e trasformato in DNA complementare (DNAc) usando un'enzima transcriptasi inversa (RT). Successivamente, il DNA si amplifica esponenzialmente attraverso la PCR utilizzando un'enzima Taq polimerasi e primers specifici per il virus. La coincidenza esatta dei primers, seguita dall'amplificazione esponenziale, finisce per produrre un test molto sensibile e molto specifico. Recentemente, sono stati creati tests basati nella PCR fluorogenica automatizzata (ossia, la RT-qPCR) per identificare il materiale genomico virale di PRRSv nei campioni clniici (figura 2). Queste PCR usano l'amplificazione in un solo passaggio in un tubo nel quale sonde fluorescenti si uniscono al prodotto della PCR man mano che il risultato avviene (ossia, PCR in tempo reale). Si crede che le PCR fluorogeniche migliorino l'affidabilità e la solidità delle RT-PCR convenzionali per identificare il PRRSv (figura 3).
Figura 3. PCR fluorogenica per identificazione del PRRSv.
Le analisi basate sulla PCR sono state realizzate con il fine di rilevare l'RNA del PRRSv nei differenti campioni clinici come: fluido orale e campioni ambientali. La PCR è particolarmente utile per l'identificazione dell'RNA virale nei campioni di seme o feci, che sono citotossici per la coltivazione cellulare o perchè non si possono valutare attraverso altre metodiche. E' anche molto utile per identificare l'RNA del PRRSv nei tessuti fetali e fluidi toracici, ossia campioni dove il virus PRRS può essere inattivato facilmente a causa dell'autolisi. L'uso della PCR è diventato comune sia per la diagnosi della PRRS come per il monitoraggio/controllo (entrata della rimonta, programmi di eradicazione o eliminazione) oppure per aumentare la biosicurezza dell'allevamento (controllo delle superfici, camion, seme, ecc).
La PCR è un test costoso rispetto ad altri metodi diagnostici. Inoltre, si deve considerare che un risultato positivo alla PCR indica la presenza dell'RNA virale, ma non necessariamente la presenza del PRRSv infettante. Inoltre la realizzazione della PCR da parte di diversi laboratori, possono variare in funzione della condizione del campione, della manipolazione del campione, della tecnica di estrazione, dei primers utilizzati, delle condizioni dei cicli termici e della capacità del tecnico che realizza il test. Pertanto, è importante che i laboratori che realizzino la PCR, convalidino i propri test, includendo stime di sensibilità, specificità rispetto agli standards di riferimento, i risultati di attitudine e di studi sperimentali o di campo.








