I metodi diagnostici più utilizzati per la diagnosi della PRRS sono l'ELISA e la PCR. In questo articolo discuteremo della PCR, il metodo con il quale si può identificare il virus in praticamente qualsiasi tipo di campione. Si basa sulla polimerizzazione del DNA ciclico di un DNA oppure, come nel caso del virus della PRRS, frammenti di cDNA affiancati da corte sequenze alle quali si uniscono i primers di DNA specifici ad ogni ciclo di PCR. La sensibilità e la specificità di un test diagnostico nel quale viene impiegata la PCR dipende di quanto specifico siano questi primers per il "target" e dai cicli termici impiegati nell'amplificazione ciclica. L'ideale sarebbe che fossero complementari solamente per il genoma di una specie virale determinata e che non fossero in grado di unirsi a nessun altro tipo di microbo o di sequenze di DNA del proprio ospite, essendo così capaci di identificare genomi di tutti i ceppi o varianti di questa specie virale. Pertanto, l'obiettivo sono i principali frammenti di genoma i più conservati.
Il virus della PRRS è uno dei patogeni per i quali è più difficile sviluppare test analitici di PCR con elevata sensibilità e specificità. I virus PRRS conosciuti oggi, appartengono a due specie: PRRSv-1 e PRRSv-2. Entrambi i virus PRRS sono diffusi a livello mondiale, ma il PRRSv-1 predomina in Europa, mentre il PRRSv-2 predomina in America ed Asia. Si può ipotizzare che la diversità genetica della specie virale dominante è maggiore di quella con minor prevalenza. Di fatto, i test in PCR utilizzati per esempio in Europa o Americhe, possono essere valutati come migliori e più idonei per la diagnosi di PRRSv-1 o PRRSv-2, rispettivamente. Di conseguenza, alcuni ceppi della specie «secondaria», o meno prevalenti, in una determinata zona geografica, potrebbero essere sottostimati per quanto riguarda la loro patogenicità nella malattia. Questo fatto è particolarmente importante per gli allevamenti dove convivono entrambi i virus PRRSv-1 e PRRSv-2. La grande diversità genetica dei virus PRRS richiede un aggiornamento costante delle sequenze dei primers e dei test PCR per poter continuare a perseguire l'evoluzione continua dei virus e parallelamente scoprire le nuove varianti genetiche completamente sconosciute. Questo è vero in particolare per il virus PRRS-1, la cui diversità genetica estrema, unito alla bassa disponibilità dei kits di riferimento per i virus in Russia, Bielorussia ed Ucraina, minaccia di compromettere la sensibilità dei kit commerciali per i subtipi genetici di PRRSv-1 dell'Europa dell'Est.

Attualmente, la variante di PCR in tempo reale è quella più utilizzata, principalmente nella diagnosi di routine. Esistono vari metodi di PCR in real time che differiscono tra loro, però quello più utilizzato è il metodo con i chiamati primers e sonde TaqMan. Una sonda TaqMan è un frammento di DNA corto (oligonucleotide) marcato con fluorofori, che si uniscono alla catena diana del DNA tra i primers collaterali. Se viene prodotta un'unione la sonda si rompe durante la polimerizzazione del DNA e lo strumento della PCR rileva la fluorescenza. La PCR in real time è quella particolarmente indicata quando si desidera una diagnosi multiplex. Questo significa che per un'unica reazione in PCR ( o in un unico test) è possibile rilevare 2 o 3 patogeni diversi attraverso l'emissione di fluorescenza con 2-3 colori diversi. Pertanto, entrambe le specie virali di PRRS possono essere diagnosticate allo stesso tempo facilmente, con la possibilità di differenziazione dei campioni di un solo soggetto, box o addirittura allevamento.
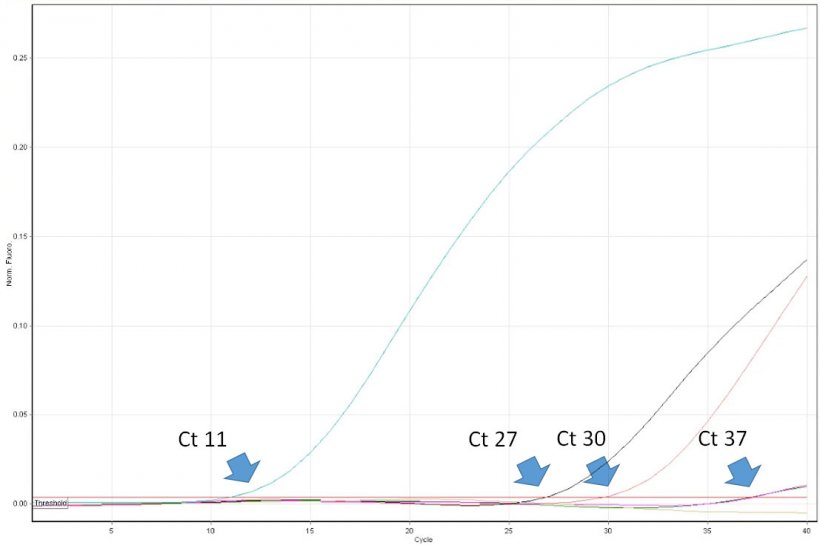
L'interpretazione dei risultati della PCR è estremamente facile e pertanto anche ingannevole. Il veterinario deve conoscere le limitazioni delle metodiche ed essere in grado di interpretare criticamente i risultati. Per esempio, un risultato positivo in PCR dimostra la presenza dell'acido nucleico diana (p. es., il virus PRRS) nel campione. Tuttavia, non dice che il campione contiene un virus infettante. La PCR in real time permette la quantificazione del virus nel campione. I risultati vengono espressi come «cicli soglie» (Ct-threshold cycle), che è il numero di volte che lo strumento di PCR ha rilevato l'emissione della fluorescenza a partire dalla reazione.Quanto minore è il numero di cicli, maggiore è il numero di coppie di DNA riscontrato (letto come numero di coppie di virus). Per esempio, un campione con Ct 20 può essere considerato altamente positivo e un campione con Ct 35 un positivo basso. Però, in alcuni casi la fluorescenza viene rilevata solamente nei cicli molto tardivi di PCR in tempo reale, per esempio quando è >38. Come possiamo interpretare valori di cicli così elevati? Purtroppo questo risultato non sempre indica la presenza di un numero molto basso di coppie nei campioni. I chiamati «virus a crescita tardiva» possono essere il risultato di una rottura spontanea della sonda dopo molti cicli termici di PCR, che simulano una lisi enzimatica del nucleotide causata dalla polimerasi DNA. Questo può succedere anche in assenza dell'acido nucleico del virus nel campione. L'importanza di un'interpretazione corretta della presenza del virus a «crescita tardiva» è massima quando si fa il monitoraggio delle popolazioni che non hanno il virus PRRS. E' necessario realizzare una diagnosi di accompagnamento, includendo i test ELISA. Sfortunatamente, alcuni laboratori d'Europa non offrono ai clienti i valori precisi di Ct in tempo reale. Al contrario, i risultati vengono espressi solamente come negativi o positivi. L'utilità di questi risultati è molto limitata per i professionisti, e in alcuni casi può dar luogo a decisioni sbagliate rispetto ai protocolli di controllo della malattia negli allevamenti.
Considerazioni pratiche
- Nessuno dei metodi attuali di PCR può rilevare tutti i tipi di virus PRRS.
- Il risultato negativo della PCR di alcuni campioni a random in un allevamento non dimostra la negatività dell'azienda.
- I risultato positivo della PCR dimostra solo la presenza dell'acido nucleico del PRRSv in un campione, ma non necessariamente la sua capacità infettante.








