Il PCV2 è comparso come una delle infezioni virali più devastanti in suinicoltura, causando un importante impatto economico sia per le perdite dirette sia per le spese per le strategie di controllo. Il PCV2 è un agente eziologico della sindrome del dimagrimento suino post svezzamento (PMWS), oggi denominata come malattia sistemica da PCV2 (MS-PCV2, figura 1). Gli studi epidemiologici e sperimentali hanno evidenziato che la diversità genetica può influenzare potenzialmente la virulenza del PVC2, caratterizzata da "ondate Globali" di genotipi emergenti e dalla circolazione di ceppi ricombinanti. Chissà se sia questa la ragione per cui la classificazione intraspecifica del PCV2 sia storicamente controversa. Nel 2008, il consorzio europeo delle malattie associate al circovirus suino ha proposto una nomenclatura standardizzata per le definizioni genotipiche del PCV2 con base nella comparazione di sequenziamenti paralleli (www.pcvd.eu). L'analisi delle sequenze dei nucleotidi del genoma del PCV2 completo e del capside (ORF2) hanno definito due vie distanziate di 0,020 e 0,035, rispettivamente. Quando la distanza (p) tra due sequenze superava questi valori, si riteneva che i ceppi appartenevano a due genotipi distinti. I risultati di queste analisi hanno riconosciuto 4 genotipi denominati: PCV2a, PCV2b, PCV2c e PCV2d (anche conosciuto come PCV2b mutante).


Figura 1. Suino di 3 mesi di vita con MS-PCV2. Notasi la colonna vertebrale marcata, indicativa di ritardo di crescita e pallore (anemia).
Dal 2008, un grande numero di sequenze di PCV2 sono state depositate nel GenBank (www.ncbi.nlm.nih.gov) e sono stati proposti vari nuovi genotipi, tuttavia, non sempre sono stati validati. Una percentuale significativa di dette sequenze, hanno un'origine ricombinante e in alcuni casi hanno ricircolato con prevalenza crescente in vari paesi asiatici e Stati Uniti. In un recente articolo (Franzo et al, 2015), una equipe internazionale di ricercatori è tornata ad analizzare la tassonomia intraspecifica del PCV2 e la definizione genotipica per comprovare la validità attuale, per unificare la nomenclatura e per evitare successivi errori di interpretazione.
I risultati ottenuti indicano che, con le sequenze oggi disponibili del capside e del genoma del PCV2, sono stati violati vari presupposti metodologici stabiliti da quasi 10 anni per stabilire gli alberi genetici. Di fatto, non è stato soddisfatto il presuposto della stesso indice di sostituzione dei nucleotidi tra i vari genotipi di PCV2. Inoltre, la definizione di un unico valore di taglio per definire i genotipi di PCV2 sembra complessa. Però, quello più importante è che gli alberi filogenetici proposti per la definizione del genotipi mancano indubbiamente di un senso logico (figura 2). Queste evidenze, assieme all'enorme quantità di nuove sequenze disponibili, implicano in modo rilevante la classificazione intraspecifica del PCV2. Pertanto, gli alberi filogenetici applicati dal 2008 per definire i genotipi di PCV2 non sono applicabili a tutti i ceppi di PCV2, per cui il metodo deve essere rivisto.
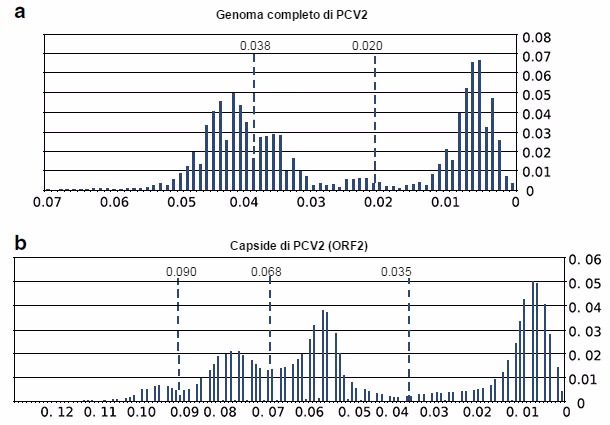
Figura 2. Risultati del confronto di sequenze del genoma completo del PCV2 (a) e delle sequenze del ORF2 (b). Sono rappresentate una percentuale di sequenze comprese in un intervallo di distanza pari a 0,01. Si indica ugualmente la via filogenetica proposta per il genoma completo (0,020), il capside (0,035), e per la via filogenetica risultante dalla nuova analisi (0,038, 0,068 e 0,090).
Come conseguenza, si propone un metodo alternativo per determinare il genotipo dei ceppi di PCV2 in modo inequivocabile. Il metodo suggerito prende in considerazione la presenza di vari ceppi ricombinanti che, di fatto, appartengono a più di un genotipo. Considerando l'elevato indice di ricombinazioni descritto, si è preferita una classificazione che tenga conto del gene ORF2. Con l'obiettivo di offrire un metodo di classificazione inequivocabile, sono state selezionate varie sequenze di riferimento, la cui classificazione era chiara secondo i metodi filogenetici. Questo ci ha permesso di definire 4 genotipi utilizzando la ricostruzione filogenetica e 47 posizioni di nucleotidi marcatori specifici per ogni genotipo. Queste posizioni dei marcatori caratterizzavano ogni genotipo in modo consistente (>95%) e potevano essere utilizzati come riferimento per assegnare una sequenza problema al determinato genotipo. Il metodo di classificzione proposto, oltre ad essere più robusto, è molto più veloce e facile da portare a termine.
La classificazione tassonomica del PCV2 continua ad essere una sfida. I risultati ottenuti confermano e convalidano la variabilità tra le sequenze virali e l'elevata frequenza delle ricombinazioni intra e inter genotipica tra i ceppi di PCV2, sottolinenado la difficoltà nello stabilire una definizione inequivocabile per questo virus. Oltre tutto, la possibilità di comparsa di nuovi genotipi di PCV2 nel futuro è molto alta e può diventare necessario avere nuovi e consistenti metodi di genotipizzazione.
La classificazione del PCV2 in genotipi è importante dal punto di vista pratico. Tutti i vaccini disponibili sul mercato europeo e nordamericano sono basati sul genotipo PCV2a, anche se i genotipi più prevalenti sono PCV2b e PCV2d. Nonostante sia già stato dimostrato un livello significativo di protezione crociata tra questi 3 genotipi, sarebbe interessante valutare l'effiacacia vaccinale nei loro confronti. Da un altro lato, fino ad oggi, nessun studio sulla filogenetica è stato pubblicato indicando che è possibile la produzione di alterazioni a livello di aminoacidi nelle sequenze di PCV2 provenienti da allevamenti vaccinati o non vaccinati. Di fatto si è speculato sulla possibilità futura che si possa avere virus mutanti dovuto alla pressione della vaccinazione, quindi il monitoraggio genotipico adeguato è fondamentale per valutare l'efficacia vaccinale.




