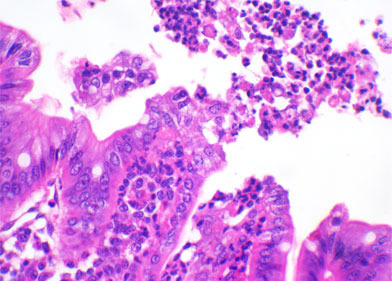
La valutazione istologica delle sezioni intestinali nominate nell'articolo precedente dovrebbero permettere il sospetto del possibile agente responsabile ed in alcuni casi definire specificatamente gli agenti eziologici. Per esempio, l'atrofia dei villi intestinali può essere causata da coccidiosi (I. suis), Rotavirus, GETv, PEDv, Deltacoronavirus o incluso C. perfringens tipo A. Se i campioni appartengono a suinetti di oltre 5-6 giorni, possiamo trovare forme sessuate o assessute di I. suis nel citoplasma degli enterociti dell'estremità delle villosità. Se non si trovano coccidi, possiamo realizzare l'immunoistochimica per rilevare la presenza di antigeni virali di Rotavirus o Coronavirus negli enterociti. In quasi tutti i casi di ETEC, l'esame istologico permetterà l'identificazione di una miriade di organismi coccobacillari adesi alla superficie degli enterociti alla base o alla parete laterale delle villosità. Il C. perfringens tipo C causerá una lesione transmurale fibrinotica ed emorragica a distribuzione segmentata nel piccolo intestino. Il C. difficile è l'unico agente infettivo che causa lesioni nel grande intestino. Spesso è presente l'edema del mesocolon. I principali risultati nei casi di infezione da C. difficile sono la riduzione delle cellule caliciformi ed una grande infiltrazione di neutrofili nella mucosa del colon, che in alcuni casi, rompono l'epitelio come un'erosione vulcanica. Il C. perfringens tipo A è più difficile da determinare attraverso l'istopatologia, dato che può determinare l'atrofia dei villi, ma il fatto di non ritrovare questo tipo di lesione non esclude questa possibile causa.
Clostridium difficile

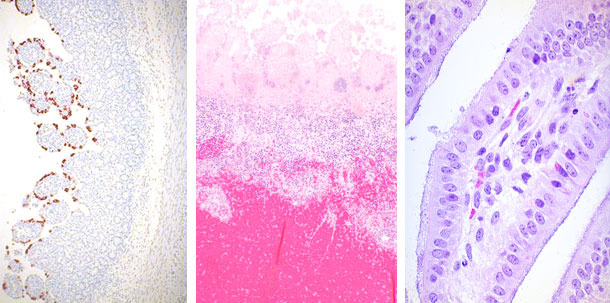
| Rotavirus | Clostridium perfringens | E. coli |
E' importante che la batteriologia dei campioni freschi dell'intestino venga realizzata sia in ambiente aerobico, sia in ambiente anaerobico. L'E. coli cresce facilmente in agar sangue e MacConkey, ma anche il semplice accrescimento di colture pure beta-emolitiche non offre una diagnosi definitiva dato che è necessaria la tipifizzazione per dimostrare la presenza di fattori patogeni, come i geni delle fimbrie (F4, F5, F41, F18,…) e tossine (LT, STa, STb, STx2e, …). Questa tipifizzazione spesso si realizza tramite PCR multiplex. Per la diagnosi di C. perfringens tipo C è fondamentale l'isolamento in anaerobiosi, assieme alla tipifizzazione molecolare (multiplex PCR) per l'identificazione dei geni delle tossine, come la beta tossina. Le routine microbiologiche per il C. perfringens tipo A e C sono identiche. Nel passato, il rilevamento del gene per la tossina beta-2 era il marcatore per i ceppi patogeni di C. perfringens tipo A, ma studi recenti del nostro gruppo e del gruppo di studio dell'Iowa State University hanno dimostrato che i ceppi beta-2 positivi di C. perfringens tipo A sono più frequenti nei suini sani rispetto a suini con diarrea. Come risultato, la definizione finale e la diagnosi di C. perfringens tipo A è la più complessa.
L'isolamento virale è difficile nella diagnosi di routine, per cui si procede con test alternativi. Quasi mai si fa la microscopia elettronica di trasmissione per l'identificazione del corona o rotavirus e solamente si realizza in alcuni laboratori americani. Con maggior frequenza, si eseguono elettroforesi in gel di poliacrilamide (PAGE) nei campioni fecali per la la verifica del RNA segmentato del rotavirus A, B e C, ma la sensibilità è molto maggiore se si usano le PCRs multiplex. Per la diagnosi di coronavirus (PED, TGE e Deltacoronavirus), nel Nord America, viene usato spesso la tecnica PCR multiplex, utilizzando una combinazione di vari primers.
Il materiale fecale del grande intestino è di molto valore per le tossine A e B di C. difficile. Siccome questo agente fa parte del microbiota intestinale normale, per la sua diagnosi è fondamentale l'identificazione delle suddette tossine medianti i kit commerciali di ELISA. E' importante dire che questi kit commerciali di ELISA sono stati sviluppati ed ottimizzati per campioni di origine umana e che la sensibilità nelle feci dei suini è bassa. Quindi la quantità del campione è importante e si consiglia di analizzare almeno da 3 a 6 campioni fecali di animali con diarrea. In base allo stesso principio, l'esame diretto delle feci si utilizza spesso per la rilevazione degli oociti di coccidi; tuttavia, siccome la sensibilità è scarsa, il consiglio è quello di raccogliere campioni di 3-5 suinetti per figliata con diarrea clinica, del 10 % delle figliate colpite con età tra 7 e 21 giorni.
Possiamo concludere che il ritrovamento degli agenti enteropatogeni nei suinetti neonati è sempre complessa. Il punto principale da non dimenticare a livello generale è che è necessario valutare tutte le possibilità. In questo senso, dobbiamo fare campionamenti ed analizzare nei confronti di tutti gli agenti potenziali causanti diarree. La PCR è uno strumento estremamente potente, ma non dobbiamo scommettere solo su questa carta. Tecniche più antiche, come l'istopatologia, possono guidarci ad interpretare i risultati ed ampliare la nostra visione.


