La diarrea è una delle principali cause di mortalità neonatale, diminuzione dell'aumento di peso e aumento dell'uso di farmaci nei suini. I principali agenti patogeni sono batteri come Escherichia coli (E. coli) e Clostridium perfringens (C. perfringens), oltre ai Rotavirus e Coronavirus.
Uno studio condotto su campioni di un Programma di Controllo in diversi paesi europei ha valutato la presenza di questi agenti patogeni nelle feci di suinetti con diarrea neonatale nel 2020.

Materiali e metodi
Tutti i campioni utilizzati in questo studio provenivano da suinetti lattanti. Un totale di 324 campioni, per lo più fecali, provenienti da 116 allevamenti sono stati testati per E. coli, C. perfringens e Clostridioides difficile (C. difficile). I campioni di 79 di questi allevamenti sono stati ulteriormente esaminati mediante PCR per i rotavirus di gruppo A e C, nonché per il virus della diarrea epidemica suina (PEDV) e per il virus della gastroenterite trasmissibile (TGEV). Pertanto, lo studio consisteva in due analisi, basate su questa differenza nello spettro dei patogeni esaminati negli allevamenti. Nella prima analisi (1) è stata valutata la frequenza dei batteri patogeni in tutti i campioni dei 116 allevamenti. Nella seconda analisi (2), i dati batterici e virali combinati su 79 allevamenti sono stati esaminati per rilevare simultaneamente più di un agente patogeno che causa la diarrea neonatale.
La maggior parte dei 116 allevamenti (figura 1) si trovava in Germania (DE; n=59), seguita da Paesi Bassi (NL; n=19) e Polonia (PL; n=19), Danimarca (DK; n= 9) , Regno Unito (UK; n=8), Austria (AU, n=1) e Irlanda (IR; n=1).

Le colture isolate di E. coli e C. perfringens in entrambe le analisi sono state tipizzate a livello molecolare per determinare la presenza di geni associati alla virulenza e quindi valutarne il potenziale virulento. (Strutzberg-Minder and Dohmann 2015). Inoltre, nell'analisi 1, il contenuto batterico semiquantitativo di E. coli, C. perfringens e C. difficile è stato analizzato in base alla loro crescita in coltura (tabella 1).
Risultati
Analisi 1:
Nell'esame batteriologico dei campioni dei 116 allevamenti sono stati trovati in totale 710 ceppi isolati (figura 2). I batteri più frequentemente isolati sono stati E. coli (48,6%), seguito da C. perfringens (33,9%) e C. difficile (15,9%). L'analisi semiquantitativa ha mostrato una crescita moderata o elevata per il 99,4% degli isolati di E. coli e il 96,7% degli isolati di C. perfringens. Tuttavia, per C. difficile è stata riscontrata una crescita moderata (46,9%) o bassa (53,1%). Solo il 16% degli isolati di E. coli aveva un potenziale emolitico (Tabella 1) e anche tra patotipi di EC definiti e EC potenzialmente virulenti (Figura 3), solo il 16% degli isolati erano emolizzanti, confermando che l'emolisi non è una caratteristica unica della virulenza.
Tabella 1: Numero (n) di isolati batterici rilevati nella coltura nell'analisi 1 e proporzioni di livelli semiquantitativi all'interno di ciascuna specie batterica.
| n | % | |||
|---|---|---|---|---|
| contenuto alto | contenuto moderato | contenuto minore | ||
| Escherichia coli | 345 | 77,1 | 22,3 | 0,6 |
| di questi, con potenziale di emolisi | 57 (16,5%) | |||
| Clostridium perfringens | 241 | 46,9 | 49,8 | 3,3 |
| Clostridioides difficile | 113 | 0,0 | 46,9 | 53,1 |
Dopo aver tipizzato gli isolati di E. coli e C. perfringens e averli collegati a ciascun allevamento, sono rimasti in totale 276 isolati di E. coli e 117 C. perfringens. Differivano l'uno dall'altro per i loro fattori di virulenza e per i loro modelli di geni delle tossine all'interno dello stesso allevamento. Sulla base del loro pattern di geni di virulenza, il 19,9% (n=55) di questi isolati di E. coli potrebbe essere assegnato ad uno dei patotipi noti associati alla diarrea neonatale (EDEC, EPEC, ETEC, NTEC). Altri 104 isolati (37,7%) erano portatori di diverse fimbrie e geni dell'adesina, nonché di tossine, quindi possono essere classificati come potenzialmente virulenti. Pertanto, un totale del 57,6% dei diversi isolati di E. coli (n=159) è stato classificato come virulento o potenzialmente virulento per la diarrea neonatale. La Figura 3 mostra la distribuzione dei risultati della tipizzazione di E. coli per paese di origine.

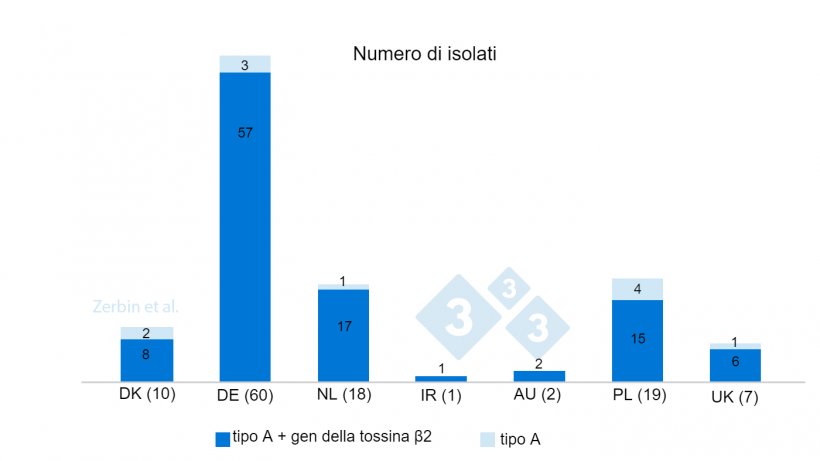
La tipizzazione degli isolati di C. perfringens ha rivelato che tutti appartenevano a C. perfringens tipo A (CPA). Il 90,6% di loro (n=106) portava i geni che codificano per entrambe le tossine α e ß2, mostrando così un alto potenziale di virulenza. Il resto delle colture isolate (9,4%, n=11) mostrava solo il gene della tossina α (FIG. 4).
Analisi 2:
Per quanto riguarda il sottogruppo dei 79 allevamenti, i patogeni riscontrati più frequentemente in questi sono stati E. coli 96,2%), C. perfringens (87,3%), Rotavirus (74,7%) e C. difficile (46,8%). Il PEDV è stato rilevato solo in due allevamenti ed il TGEV in nessuno. Pertanto, il gruppo Coronavirus non è stato considerato nell'analisi successiva.
La successiva tipizzazione ha rivelato isolati di E. coli virulenti o potenzialmente virulenti nel 78,9% (n=60) degli allevamenti e CPA con il gene della tossina ß2 nell'81,0% (n=64).
Nel 25,3% (n=20) degli allevamenti sono stati trovati contemporaneamente quattro diversi patogeni, tre diversi patogeni nel 39,2% (n=31), due patogeni nel 31,6% (n=25) e nel 3,8% (n=3) degli allevamenti è stato rilevato un solo patogeno. Solo in uno degli allevamenti non è stato trovato nessuno dei patogeni analizzati.

Tra questi, la combinazione di E. coli con Rotavirus e CPA con tossina β2 si è verificata nella maggior parte di questi allevamenti (n=19/24,1%). L'associazione con E. coli, CPA e C. difficile si è verificata significativamente meno frequentemente (n=8).
L'E. coli è stato l'agente patogeno più frequentemente riscontrato, principalmente in combinazione con CPA con il gene della tossina β2 (64,6%) o con rotavirus (64,6%).
Discussione
Il presente studio ha analizzato la presenza di agenti patogeni rilevanti che causano diarrea neonatale negli allevamenti europei che hanno partecipato volontariamente ad un programma di monitoraggio della diarrea dei suinetti. L'interpretazione dei risultati attuali deve essere eseguita con cura, poiché vi è una certa distorsione di selezione nel disegno del campione. Gli allevamenti hanno inviato i campioni volontariamente, poiché presentavano problemi di diarrea. Tuttavia, i dati mostrano tendenze realistiche e chiare in relazione ai patogeni che causano la diarrea neonatale che sono già state descritte in letteratura.
Le due analisi in questo studio confermano che E. coli e C. perfringens sono i principali patogeni batterici che causano la diarrea nei suinetti lattanti. I dati attuali hanno mostrato che quasi il 58% degli isolati di E. coli e quasi il 91% degli isolati di C. perfringens possono essere considerati virulenti o potenzialmente virulenti. Nella seconda analisi, sono stati trovati isolati di E. coli e C. perfringens in circa l'80% degli allevamenti.
Un risultato interessante è la bassissima evidenza di Coronavirus (PEDV e TGEV) nei campioni di studio. Sebbene siano citati come possibili agenti di diarrea neonatale, sono scarsamente descritti in letteratura e la diarrea causata dal TGEV è diventata rara in Europa. L'analisi combinata dei diversi agenti patogeni batterici e virali nei 79 allevamenti ha rivelato la presenza simultanea di due o più agenti patogeni in quasi il 95% degli allevamenti. Ciò dimostra che la diarrea neonatale nei suinetti ha raramente un'eziologia monocausale. Questo dovrebbe essere considerato quando si prendono decisioni riguardanti misure profilattiche e terapeutiche.