Le sfide, non così nuove, quando si parla del virus della PRRS
Non è una novità che il virus della PRRS abbia caratteristiche importanti che contribuiscono alla costante ricomparsa della malattia negli allevamenti di suini, causando notevoli problemi sanitari agli animali e perdite economiche. Una di queste caratteristiche è la sua rapida capacità di mutare e potenzialmente diventare più grave e/o addirittura infettare animali precedentemente “immuni” a diverse varianti della PRRS. Per questo motivo, per conoscere la “tipologia” di vPRRS presente in un allevamento – se si tratta del virus di una nuova epidemia o di un virus “residente” – viene spesso analizzato il genoma del vPRRS.
Questo compito viene generalmente eseguito sequenziando una parte importante del vPRRS chiamata gene ORF5; per capire dove si inserisce il virus dal punto di vista evolutivo e come si confronta con i virus precedentemente rilevati nell’allevamento o nelle vicinanze/regione.

Le informazioni fornite dal sequenziamento del vPRRS sono state ampiamente utilizzate sul campo per:
- capire quale virus sta causando problemi
- per conoscere l’origine dell’epidemia
- per distinguere tra ceppi vaccinali e ceppi “residenti”/nuova introduzione
- per la scelta dei vaccini, ecc...
Recentemente, la classificazione per “lineage” è stata sempre più utilizzata per classificare i virus. Questo metodo è stato sviluppato intorno al 2010 (Shi et al., 2010), quando i vPRRS erano divisi in 9 linee distinte; e divenne ancora più preciso quando furono suddivisi in sub linee.
Una verità scomoda: è sufficiente sequenziare un campione?
Il sequenziamento del virus della PRRS di solito non è economico, quindi viene generalmente eseguito in “situazioni speciali” e non di routine (ad esempio durante nuove epidemie, dopo casi di inoculazione del virus, ecc.). Inoltre, in queste situazioni, nella stragrande maggioranza dei casi studiati per la diagnosi, per il sequenziamento viene utilizzato un solo campione o pool di campioni, tra i tanti che solitamente vengono prelevati in allevamento.
L’impatto è che, in molti casi, i veterinari, gli allevatori e altro personale legato alla salute degli animali, sono soliti utilizzare le informazioni di una singola sequenza per trarre conclusioni importanti sull’origine del virus (soprattutto in caso di nuove epidemie), orientare le indagini sulle epidemie e informare sugli interventi futuri.
Nel nostro studio, volevamo confermare che, se sequenziassimo più campioni raccolti nello stesso giorno, tutti fornirebbero la stessa sequenza, o sequenze che sarebbero “abbastanza vicine” da essere considerate almeno appartenenti alla stessa linea virale. Volevamo anche provare il sequenziamento dell'intero genoma (WGS, whole-genome sequencing, in sigla inglese) insieme al sequenziamento ORF5 per vedere se potevamo ottenere maggiori informazioni. Il WGS potrebbe avere il vantaggio di fornire informazioni sull’intero virus, mentre tradizionalmente veniva utilizzato l'ORF5, ma ci racconta solo una parte della storia. Tuttavia, poiché è uno sviluppo ancora relativamente recente e può essere costoso (negli Stati Uniti costa circa tre volte di più del sequenziamento ORF5), stiamo ancora imparando come interpretarlo e il nostro “database” per il confronto è molto più piccolo rispetto a quello di ORF5.
Sospettavamo che, dato il rapido tasso di mutazione della PRRS e le grandi dimensioni dei moderni allevamenti di suini, avremmo potuto trovare più linee di virus della PRRS all'interno di un evento di campionamento, il che sarebbe stato preoccupante. Una sintesi grafica del ragionamento alla base del nostro studio è mostrata in Fig. 1.
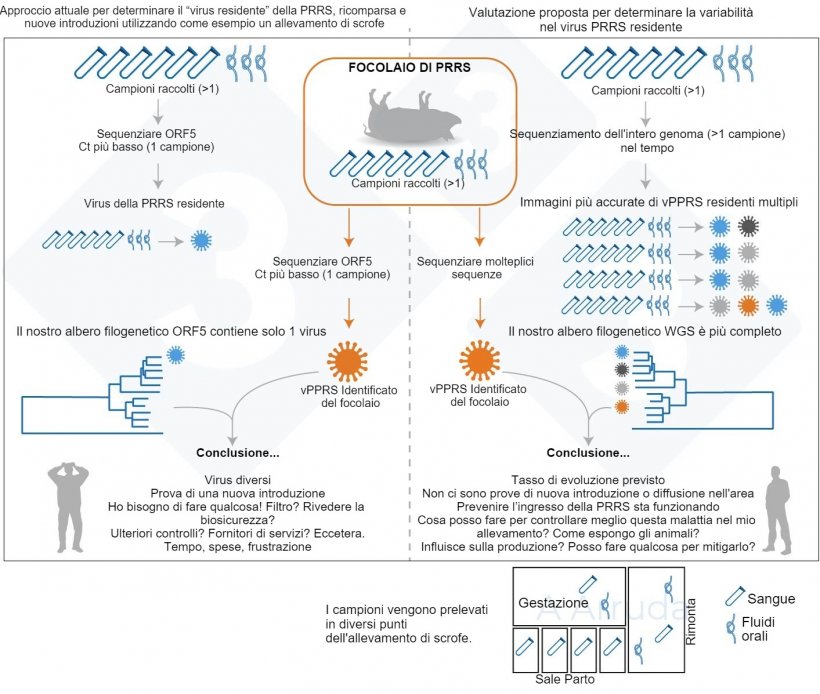
Investigando su quanti ceppi di virus della PRRS possiamo trovare nei nostri allevamenti
Includiamo 5 allevamenti nello studio, 3 scrofaie e 2 allevamenti d'ingrasso; e abbiamo raccolto campioni mensilmente per circa un anno utilizzando diversi tipi di campioni (raschiature tonsillari, fluidi orali, emosieri). Abbiamo sempre campionato le stesse posizioni del capannone distribuite spazialmente in tutta la struttura per cercare di ottenere la massima rappresentazione possibile dell'intero sito. Abbiamo raccolto fino a 16 campioni per sito ogni mese e tutti sono stati analizzati utilizzando la PCR quantitativa; l'ORF5 è stato quindi sequenziato nei campioni positivi.
Cosa abbiamo scoperto?
La nostra scoperta più importante è stata che, in condizioni di campo, siamo stati in grado di rilevare fino a 3 diversi linee (lineages) del vPRRS durante un singolo evento di campionamento nelle scrofaie e fino a 2 negli allevamenti di ingrasso (Figura 2).
Figura 2. Sub-linee del vPRRS riscontrati negli allevamenti oggetto dello studio (1-5) durante la durata dei nostri eventi di campionamento (in mesi) per i diversi tipi di campione.
| Evento di campionamento | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Allev. | Tipo di campione | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| 1 | Emosieri | L1H | L1H | ||||||||||
| Raschiatura di tonsille | |||||||||||||
| 2 | Emosieri (Processing fluid) | L1H | L1H | L1A | L1H | L1H | |||||||
| L1H | |||||||||||||
| L8† | |||||||||||||
| Raschiatura di tonsille | L1H | ||||||||||||
| 3 | Emosieri | L1H | L1H | L1H | L1H | ||||||||
| Raschiatura di tonsille | |||||||||||||
| 4 | Fluidi Orali | L5† | L1A | ||||||||||
| L5† | |||||||||||||
| Raschiatura di tonsille | L1A | L5† | L5† | L1A | |||||||||
| L5† | |||||||||||||
| 5 | Fluidi Orali | L1A | L1A | L5† | |||||||||
| Raschiatura di tonsille | L1A | L1A | |||||||||||
Abbiamo anche osservato che, nel caso del nostro studio, i campioni di fluido orale erano molto difficili da sequenziare, mentre il trattamento dei campioni di emosieri era il più semplice. Considerando che entrambi sono campioni “composti”, ciò potrebbe essere spiegato dai valori Ct più elevati, che indicano una minore quantità di virus nei fluidi orali rispetto agli emosieri (processing fluids). Abbiamo tentato di eseguire il WGS, ma alla fine non siamo riusciti a ottenere sequenze complete dell'intero genoma per nessuno dei nostri campioni. Sospettiamo che ciò possa essere dovuto al tipo di campioni utilizzati, ma sono necessarie ulteriori ricerche su questo argomento.
Questi risultati dimostrano quanto sia importante considerare il sequenziamento di più campioni quando si prendono decisioni importanti, anche se comprendiamo che ciò comporterà costi più elevati.
Ringraziamenti
Il National Pork Board ha finanziato questo progetto. Vorremmo ringraziare i veterinari e gli allevatori che sono stati determinanti nella raccolta dei campioni...






