Nonostante la PRRS sia una malattia relativamente nuova, comparsa per la prima volta in Canada nel 1979, ora è endemica in quasi tutti i paesi del mondo, e causa danni economici devastanti dovuto alla riduzione della produzione di suinetti, problemi riproduttivi e perdite di performance dei suini. I calcoli suggeriscono che da 1/4 ad 1/3 dei danni economici causati da malattie siano attribuibili alla PRRS. La malattia è di tipo persistente, in modo che gli animali vanno incontro al recupero ma allo stesso tempo eliminano e continuano ad infettare altri soggetti. Questo quadro suggerisce una produzione tardiva di anticorpi neutralizzanti, una insufficiente risposta immunitaria sia innata che acquisita, ed una mutazione da parte del virus durante l'attività di replicazione, aumentando la sua diversità genetica.
Un Comitato Internazionale coordinato sta studiando i vari aspetti della malattia, ma il “santo gral” nel produrre suini geneticamente resistenti è ancora lontano. Senza dubbio, gli strumenti ed il know how sviluppati dai progetti ‘genoma’ stanno offrendo nuove opportunità per la conoscenza dei componenti genetici della risposta all'infezione dal virus PRRS.
Esistono molte evidenze di una variabilità significativa della risposta alla tolleranza tra razze. Per esempio, studi tedeschi hanno dimostrato che dopo l'esposizione alla PRRS, una linea di suini nani presentava una riduzione sia della durata che della persistenza del virus; anche la carica virale era considerevolmente più bassa rispetto ad una linea commerciale come la Pietrain. Negli Stati Uniti, sono state dimostrate differenze significative delle risposte di suini Duroc-Hampshire rispetto a suini Large White-Landrace.
Uno studio collaborativo dell'Istituto Roslin di Edimburgo fu il primo a studiare l' SNP (Un polimorfismo a singolo nucleotide, spesso definito in inglese Single Nucleotide Polymorphism o SNP, pronunciato snip): è un polimorfismo, cioè una variazione, del materiale genico a carico di un unico nucleotide, tale per cui l'allele polimorfico risulta presente nella popolazione in una proporzione superiore all'1%. Al di sotto di tale soglia si è soliti parlare di mutazione di tutto il genoma.Spesso si è chiesto: “ si può determinare una parte della variabilità genetica attraverso marcatori genetici?”

I ricercatori hanno analizzato i dati accumulati di oltre 8000 figliate in un solo allevamento nel periodo di 10 anni durante i quali sono stati registrati gli episodi di PRRS. Mediante un chip SNP di 7K sono stati identificati vari SNP significanti ed indipendenti, il che suggerisce che esistono molte regioni genetiche che regolano la tolleranza al virus PRRS. I genotipi studiati erano: Landrace, Large White, Pietrain, Meishan, Duroc e vari ibridi. Curiosamente, l'impatto del virus fu maggiore nella razza Meishan che nei suoi equivalenti europei. In altri allevamenti si sta portando a termine uno studio utilizzando un chip SNP più ampio ed attuale per vedere se gli SNP identificati continuano ad essere importanti negli allevamenti e anche per osservare la presenza di effetti adversi correlati con le caratteristiche produttive degli animali.
Nel frattempo anche in Italia sono stati raccolti dati che combinano i campioni di DNA con dati genealogici e di produttività con dati sanitari e di produzione. Per questo sono stati fatti oltre 5000 campioni individuali di 4 linee genetiche (Duroc, Landrace, Large White e Pietrain) di 18 allevamenti geneticamenti correlati. Sono stati identificati vari SNP molto signficativi, ed il lavoro continua ricercando marcatori e se questi sono consistenti lungo il periodo di prova.
Rappresentazione in scala multidimensionale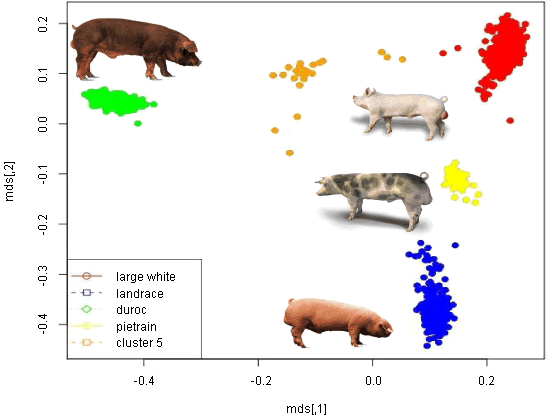 Relazione genomica – Scala multidimensionale (MDS): distribuzione dei clusters SNP per razze. |
I risultati delle ricerche finora indicano che ci sono variabilità genetiche sufficienti nella risposta all'infezione per poter sperare miglioramenti selettivi: quindi ci sono motivi per credere che sia possibile produrre animali commerciali tolleranti alla malattia attraverso la selezione. Senza dubbio , la complessità della malattia e l'elevato numero di markers potenzialmente nascosti suggerisce che la redditività della produzione di suini in grande quantità da linee genetiche resistenti non potrà verificarsi in brevissimo tempo.
Definizioni:
Un chip SNP è una piccola particella di silicone che contiene molte sonde di DNA. Queste sonde si legano selettivamente a molecole di Dna la cui sequenza di aminoacidi siano totalmente complementari - ad ogni T corrisponde una A, e ad ogni G corrisponde una C. Questi chip possono essere utilizzati per identificare sequenze specifiche di DNA e per cercare sequenze di DNA che differiscono per il "polimorfismo nucleotidico semplice".
SNP (si pronuncia SNIP) vuol dire ‘polimorfismo a nucleotide semplice". E' la differenza di un solo aminoacido tra i suini singoli nel codice genetico, o sequenza di DNA. Gli SNP sono le variazioni genetiche più frequenti tra soggetti.





