L'Escherichia coli è parte integrante della flora intestinale dei suini ed in determinate circostanze causa quadri clinici di colibacillosi, malattia che si può manifestare in varie modalità come diarrea neonatale, diarrea post-svezzamento, malattia degli edemi, setticemia, ecc..
Una diagnosi adeguata comprende un'anamnesi completa, un corretto campionamento ed una diagnosi differenziale supportato da diverse tecniche. Il semplice ritrovamento di un ceppo patogeno non sempre giustifica la malattia, dato che è comune trovare portatori asintomatici. L'identificazione dei ceppi rilevanti per la loro virulenza e la loro differenziazione dal resto della flora abituale dell'intestino è un tipo di diagnosi moderna e la PCR in tempo reale (qPCR) applicata all'identificazione dei fattori di virulenza (FV) dell'E. coli, si presenta come un potente strumento capace di dare informazione di ottima qualità.

A partire dall'estrazione degli acidi nucleici di differenti matrici biologiche (coltivazione, feci, intestino o tampone rettale), i test qPCR riconoscono regioni specifiche del genoma dell'E. coli che codificano distinti FV. In questo studio, ogni prova fu disegnata per l'identificazione di ogni FV (vedere tabella 1). Una qPCR aggiuntiva (ECCO) fu utilizzata per confermare la presenza di E. coli nei campioni ed assicurare che tutte le tappe siano state realizzate correttamente. La qPCR ha un carattere qualitativo, ma anche quantitativo (Immagine 1), vantaggio importantissimo rispetto alla PCR tradizionale.
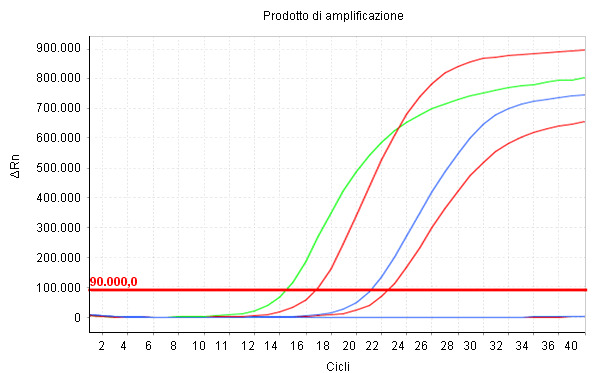
Immagine 1. I risultati dell'amplificazione del campione vengono esposti in tempo reale man mano che i cicli termici avanzano in modo che il livello di fluorescenza registrata (ΔRn) aumenta esponenzialmente. Il Cq è il numero dei cicli nei quali la fluorescenza inizia ad essere rilevata. Quanto minore è il valore di Cq, maggiore è la concentrazione del parametro studiato nel campione.
La valutazione dei ceppi virulenti nei processi di colibacillosi realizzata finora, passa attraverso la coltivazione microbiologica iniziale, con l'isolamento delle colonie di E. coli e successiva caratterizzazione dei FV. Questa metodica presenta lo svantaggio di essere un'analisi limitata al numero di isolamenti selezionati avendo fiducia della precisione della diagnosi e della rappresentatività di questi nella popolazione complessiva di E. coli nel campione in studio. Così, si corre il rischio di passare inavvertitamente a popolazioni di E. coli non maggioritarie che magari non hanno importanza clinica.
La qPCR offre la possibilità di realizzare l'analisi dei FV sul campione clinico. Questa metodica evita tappe intermedie di coltivazione e le rispettive determinazioni su ognuno dei differenti isolati selezionati, il che porta ad un impiego inferiore di tempo e denaro. Dato il carattere quantitativo della tecnica si può stabilire una relazione tra il numero di coppie rilevate per un determinato gene di virulenza e la popolazione totale di E. coli presente nel campione. In questa maniera è possibile interpretare la probabilità di successo di isolamento di un ceppo con una combinazione specifica per FV.
Questa metodica presenta certe limitazioni a causa del numero indeterminato di coppie dei vari geni che codificano ogni FV all'interno di differenti popolazioni batteriche. Nonostante ciò, soddisfa l'obiettivo proposto: valutare la possibilità di trovare in un campione una popolazione di E. coli con una specifica combinazione di FV.
Qui di seguito abbiamo un esempio pratico. Di fronte ad un sospetto clinico di malattia degli edemi in suini all'ingrasso, sono stati spediti in laboratorio 4 intestini piccoli (IP) appartenenti a soli soggetti colpiti per valutare la presenza di E. coli verotossigenico. Il pool formato dai 4 intestini furono analizzati mediante qPCR, portando al risultato dei valori Cq per F18, Stx2e e ECCO quasi coincidenti (Tabella 1). Questi risultati possono essere interpretati come la presenza di una popolazione ampiamente maggioritaria di E. coli che possiede i geni codificanti per F18 e Stx2e, ossia, un ceppo verotossigenico. Inoltre, vi è stato un valore debolmente positivo per F4 (Cq=36,9) il che indica una presenza molto minoritaria di popolazione per questo FV.
Allo stesso tempo, è stata realizzata una coltivazione microbiologica dei campioni nella quale 12 colonie di E. coli furono selezionate. Il confronto fenotipico ha rivelato la presenza di un unico ceppo (Immagine 2). La caratterizzazione della stessa attraverso i suoi FV con la qPCR confermò che si trattava di un ceppo potenzialmente verotossigenico, dato che furono identificati simultaneamente i geni codificanti per F18 e Stx2e.
Tabella 1. Risultati qPCR.
| FV | Pool 4 piccoli intestini | Ceppo E. coli isolato |
| F4 | POS (Cq = 36,9) | neg |
| F5 | neg | neg |
| F6 | neg | neg |
| F18 | POS (Cq = 22,28) | POS (Cq = 16,58) |
| F41 | neg | neg |
| LT | neg | neg |
| STa | neg | neg |
| STb | neg | neg |
| Stx2e | POS (Cq = 22,39) | POS (Cq = 17,29) |
| eae | neg | neg |
| ECCO | POS (Cq = 21,1) | POS (Cq = 16,97) |
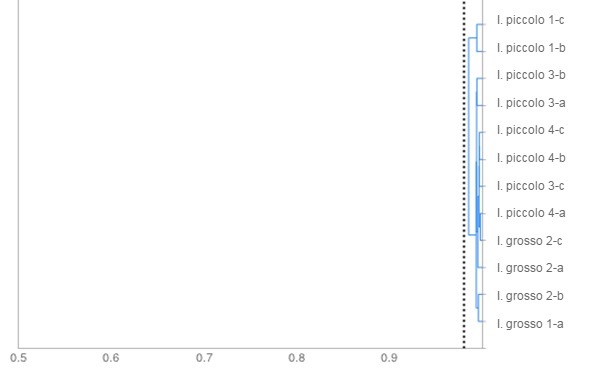

Immagine 2. Dendogramma. Si evidenzia il grado di similitudine tra gli isolati di E. coli in funzione delle caratteristiche di fermentazione degli zuccheri. Tutti gli isolati hanno una similitudine superiore alla soglia di taglio stabilita in 0,98 (linea punteggiata); il che ci fa concludere che si tratta di un unico ceppo.
In conclusione, la qPCR, applicata direttamente sui campioni clinici, può rappresentare una economia sostanziale di risorse e di tempo offrendo informazioni capaci di permettere un'interpretazione che porti alla soluzione di una delicata diagnosi di colibacillosi suina.




